Mesure de la similarité entres molécules
Introduction
Pour évaluer la similarité entre 2 molecules A and B ,il faut d'abord définir un schéma déscriptif commun à toutes les molécules quelque soit leur taille,type d'atomes,type de liaisons. La génération automatique de descripteurs (qsar) permettra par exemple de retenir comme schéma déscriptif de chaque molécule, le Logp,Moment dipolaire,Ehomo,..... Chaque molécule est donc représentée dans un espace multidimensionnel par un vecteur comprenant la valeur de chaque déscripteur. La méthode la plus utilisée pour comparer 2 molécules est de créer un chromosome ou la valeur d'un géne signifie la presence ou l'absence d'un descripteur particulier.

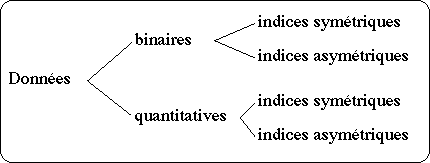
Les Indices de similarité
Lorsqu'on compare 2 objets ayant pour un descripteur donné la valeur "zéro" ,ce double zéro pose le probléme de la ressemblance. C'est la raison pour laquelle on distingue deux classes d'indices d'association. les indices qui considèrent le double zéro comme une ressemblance (au même titre que d'autres identités de valeurs) sont dits symétriques, les autres asymétriques.
Le choix d'un indice approprié est fondamental, car toute analyse ultérieure se fera sur la matrice d'association qui en résulte.
 1)Indices de similarité binaires symétriques
1)Indices de similarité binaires symétriques
Les indices de cette catégorie :
![]() travaillent sur des données binaires
travaillent sur des données binaires
![]() traitent un double zéro de la même manière qu'un double 1.
traitent un double zéro de la même manière qu'un double 1.
Le plus typique représentant de cette catégorie est l'indice de simple concordance S1 Il se construit de la manière suivante:

 2)Indices de similarité binaires asymétriques
2)Indices de similarité binaires asymétriques
Cette catégorie forme le pendant de la précédente et est destinée à comparer des objets sur la base de présence-absence de descripteurs. Les formules sont du même type que ci-dessus, mais ne font pas intervenir le cas des doubles zéros. Les indices les plus connus sont celui de Jaccard (S7) et celui de Sørensen (S8):

 3)Indices de similarité quantitatifs symétriques
3)Indices de similarité quantitatifs symétriques
L'indice fait le rapport entre le nombre de descripteurs ayant le même état
pour les deux objets et le nombre total de descripteurs.
D'autres indices de cette catégorie sont intéressants parce qu'ils permettent
de comparer au sein d'un même coefficient des descripteurs de types mathématiques
différents.
On calcule pour chaque paire d'objets,des similarités partielles pour chaque descripteur,
puis de faire la moyenne des similarités obtenues.
Parmi les indices de ce genre, citons l'indice d'Estabrook & Rogers (S16)
et l'indice de Gower (S15).
 4)Indices de similarité quantitatifs asymétriques
4)Indices de similarité quantitatifs asymétriques
Cette catégorie, destinée aux données d'abondances d'espèces, recèle plusieurs indices fréquemment utilisés. Mentionnons-en deux: l'indice de Steinhaus S17, et la similarité du khi-carré, S21. Le S17, pour deux objets, compare pour chaque espèce la plus petite des abondances à la moyenne des deux objets:

 Indice de Tanimoto
Indice de Tanimoto
Le coefficient de Tanimoto T est défini comme le rapport

Ou A(i) et B(i) sont égaux à 1 quant le i-iéme descripteur est trouvé dans la molecule A et B respectively et 0 quant il est absent; M is le nombre total de descripteurs dans la base. T apaprtient à la seconde classe de coefficients d'association definis ci dessus.
 Indice de Tversky
Indice de Tversky
Mesure de distance entres molécules
Mesures de distance pour descripteurs qualitatifs ou binaires
Tous les coefficients de similarité peuvent être transformés en distances par l'un ou l'autre des
procédés suivants:

Mesures de distance pour descripteurs quantitatifs
Au contraire des mesures de similarité, les mesures de distance accordent une valeur maximale à deux objets
complètement différents et minimale (0) à deux objets identiques.
On distingue deux catégories d'indices de distance selon leurs propriétés géométriques:
- les métriques, qui obéissent aux quatre propriétés suivantes:
1. Si a = b, alors D(a,b) = 0
2. Si a différent de b, alors D(a,b) > 0
3. D(a,b) = D(b,a)
4. D(a,b) + D(b,c) est plus grand ou égal à D(a,c)
- les semi-métriques, qui n'obéissent pas à la quatrième propriété ci-dessus,
et qu'on ne peut donc positionner correctement dans un espace euclidien
.
Le plus courant des indices de distance métriques est la distance euclidienne (D1).
Citons également;
 .Distance de Manhattan
.Distance de Manhattan
 .Distance de Levenhstein
Dans D1,chaque descripteur y est considéré comme une dimension dans un espace euclidien,
les objets sont positionnés dans cet espace en fonction de la valeur prise par chaque descripteur,
et la distance euclidienne entre 2 molécules x1 et x2 se mesure selon la formule :
.Distance de Levenhstein
Dans D1,chaque descripteur y est considéré comme une dimension dans un espace euclidien,
les objets sont positionnés dans cet espace en fonction de la valeur prise par chaque descripteur,
et la distance euclidienne entre 2 molécules x1 et x2 se mesure selon la formule :
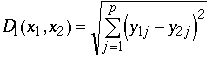
Cette mesure n'a pas de borne supérieure. De plus, ses valeurs s'accroissent avec le nombre de descripteurs, et surtout la distance varie avec l'échelle de chacun des descripteurs. C'est pour cette dernière raison qu'on calcule le plus fréquemment la distance euclidienne après centrage et réduction des variables. Cette pratique est aussi courante en analyse en composantes principales,en analyse Kohonen qui préserve la distance euclidienne entre objets. Plusieurs variantes de D1 ont été proposées pour pallier à l'un ou l'autre de ses inconvénients. Citons la distance de corde, qui calcule une distance euclidienne entre objets après avoir normé les vecteurs-objets à 1. Cette astuce rend D3 insensible aux doubles zéros, et appropriée aux données d'abondances d'espèces. Enfin, parmi les coefficients de distance semi-métriques, le plus utilisé est D14, la distance de Bray & Curtis, qui est la réciproque de la similarité de Steinhaus: D14 = 1-S17.
