CADCOM/MANUEL D'UTILISATION
LE POTENTIEL DE LIPOPHILIE MOLECULAIRE
1) Définition du PLM
Les méthodes traditionnelles donnent une valeur 1D du Logp
en ce sens qu'elles fournissent des informations limitées
et insuffisantes lorsqu'il s'agit d'analyser la structure
dans l'espace et notamment d'étudier les interactions
moléculaires.
Le potentiel de lipophilie moléculaire de DUBOST et KUMMER
est une approche originale permettant de définir des zones
lipophiles et hydrophiles dans l'espace en associant le
potentiel de lipophilie à la notion de probabilité de
présence de ce potentiel.
Si on considére une molécule S comme un ensemble
de n fragments indépendants i,il est possible de
quantifier la lipophilie de ces différents fragments
au moyen de constantes hydrophobes fi.
Lorsque cette molécule est plaçée au sein d'une phase
constituée de molécules apolaires ou peu polaires L,
leur disposition aléatoire à une certaine distance
relativement importante de S va se trouver modifiée
au fur et à mesure que cette distance diminue.
En effet,les fragments lipophiles constitutifs de S
ont tendance à attirer les molécules L alors que
les fragments hydrophiles les repoussent.L'arrangement
des molécules L autour de S et l'apparition des
liaisons intermoléculaires de type hydrophobe sont
donc fonction des différentes valeurs des constantes
fragmentales fi et des distances di ,exprimées en Ang,
séparant L de chaque fragment i.
Celà peut se traduire de façon quantitative en
définissant ,en tout point M entourant S ,un potentiel
de lipophilie moléculaire(PLM) de la façon suivante:
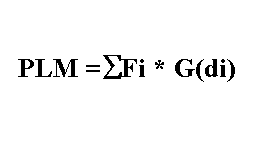 ou G(di) est une fonction directe de la distance de M
à chaque atome.
ou G(di) est une fonction directe de la distance de M
à chaque atome.


Webmaster:
Last revised:05/2000